
PacBio®シークエンシングシステム

PacBio®ロングリードのコアテクノロジー
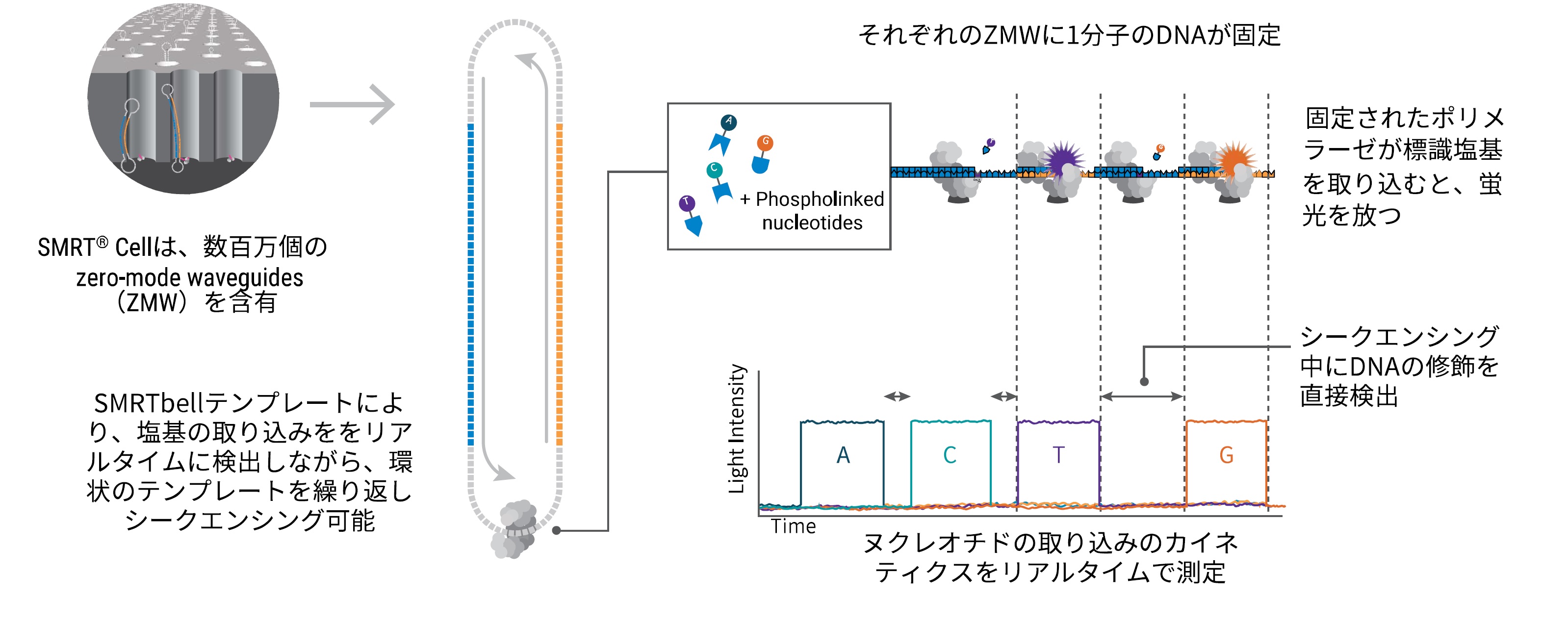
PacBio®のロングリードシークエンシングテクノロジーは、1分子リアルタイム(SMRT®)シークエンシングテクノロジーを基盤としています。
この革新的なアプローチは、DNA複製という自然なプロセスを利用して、長い断片のネイティブなDNAをシークエンシングし、何百万ものウェルから同時にデータを取得することを可能にします。
PacBio®ロングリードシークエンシングの仕組み
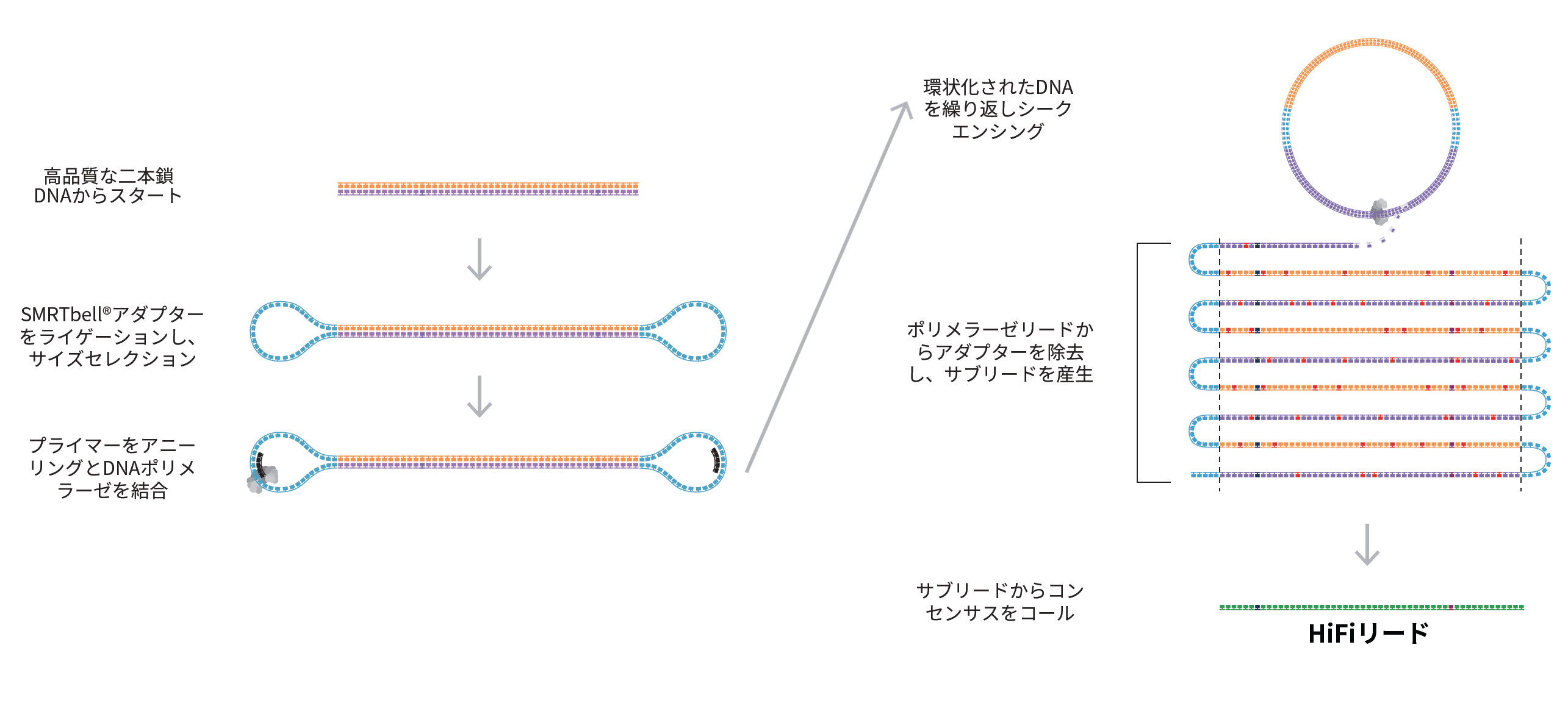
HiFiリード
Circular Consensus Sequencing (CCS) を用いて99.9%以上の精度を持つHiFiリードを生成し、生物学上の難問に答えるための最も有益なデータにアクセスできます。
PacBio®ショートリードのコアテクノロジー
PacBio®ショートリードシステムは、高精度の読み取りを実現することに焦点を当てたシークエンサーです。独自のSBB®(Sequencing by Binding)ケミストリーは、ネイティブヌクレオチド、スカーレスインクルージョン、結合と伸長のための最適化された条件を使用しています。これらの技術革新により、90%以上のリードがQ40という極めて精度の高いデータが得られます。
シークエンシングシステム
PacBio®認証サービスプロバイダー

PacBio®認証サービスプロバイダーは、SMRT®シークエンスを用いたサービスを展開する受託サービス機関です。厳格な技術トレーニングとベストプラクティスを学んだパートナーが、高品質な結果を提供します。
Pacific Biosciences社が提供するDNAシークエンサーおよび試薬は研究目的のみに使用できます。
診断目的およびその手続き上での使用はできません。
お問い合わせ
- お気軽にお問い合わせください。
- TEL:03-6240-0843







