次世代シーケンスデータ解析ソフトウェアStrand NGS

Strand NGS最新情報INFORMATION
特徴
Strand NGSは、次世代シーケンスデータの解析、マネジメント、図示化ができる統合的なツールです。RNA-Seq, DNA-Seq, ChIP-Seq, Methyl-Seq, small RNA-Seq, MeDIP-Seqに対応しています。
スタンダードな生物種のシーケンスアノテーションを簡単にダウンロードでき、その他の生物種についてもアノテーションを作成することが可能です。
多様なQuality Controlとフィルタリングで品質の低いデータを解析から除外します。GeneOntology、GSEA、Pathway解析などで生物学的解釈を調べられるので、単なるリード情報を実験のゴールに結び付けることができます。
対応アプリケーション
製品概要
アライメント
Strand NGSにはBurrows Wheeler Transformに基づいた、独自のアラインナーが搭載されています。Illumina, Ion Torrent, ABI, 454 (Roche), Pac Bio由来のsmall RNAのリードとDNAリード(ChIP-Seq、DNA-Seq、MeDIP-Seq)と、RNAのリード(スプライス、スプライス無しどちらも可)のアラインメントができます。通常アラインメントアルゴリズムはリードやアラインメントの特性に応じて制限がありますが、Strand NGSのアラインナーはロングリード、ショートリードどちらにも対応しています。ギャップやミスマッチを許容し、シングルエンド、ペアエンドどちらもアライン可能です。
※Methyl-Seqデータのアラインメントはできません。
数値化とノーマライズ
遺伝子レベル、エキソンレベル、トランスクリプトレベルでの発現値。
ノーマライズ方法としてDESeq、Quantile、TMM、Normalize Total Sample Read Count。
データのQuality Control
アラインメント前後のQC、低品質のリードのフィルター、アダプターの除去。
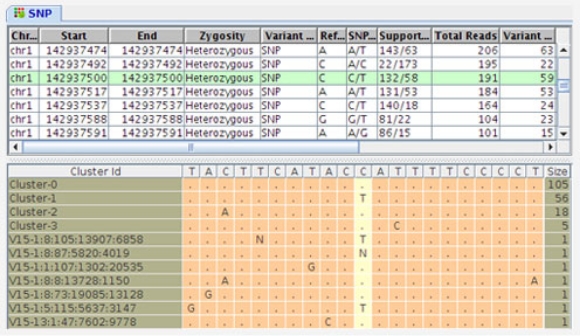
データのビジュアル化
Gene View, Variant Support View, Elastic Genome Browser
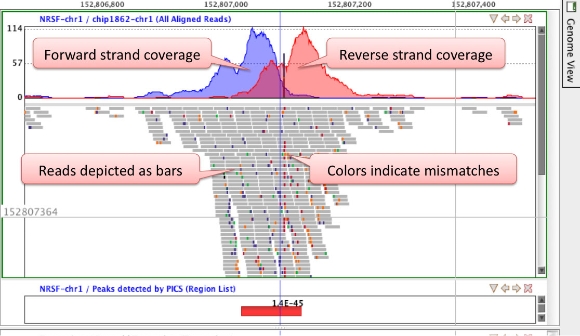
Genome Browser
ゲノム上にどのようなリードがあるかを可視化。
ピーク領域、SNP、融合遺伝子などの様々な解析結果と、Cytoband、遺伝子、転写産物などのアノテーションデータを重ね合わせることが可能です。
複数の異なる領域を同時に見たり、ズームしたり、ブックマークしたりもできます。
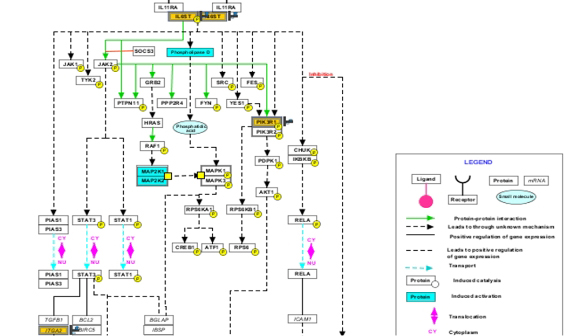
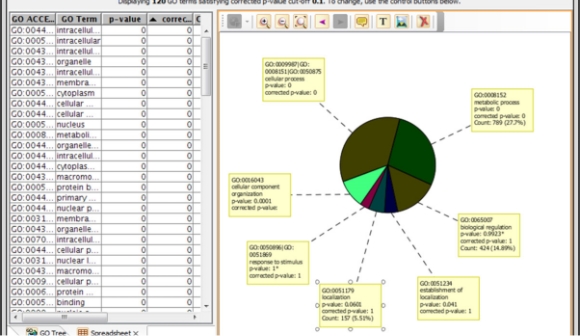
Biological Interpretation
既知のPathwayを使用したPathway解析、NLP由来のネットワーク作成、GeneOntology、GSEA、GSA解析。
生物種
| 脊椎動物 | ヒト、マウス、ラット、ゼブラフィッシュ、ヒツジ、ブタ、ウマ、アカゲザル、オポッサム、ニワトリ チンパンジー、ウシ、マーモセット、キンカチョウ |
|---|---|
| 昆虫 | ショウジョウバエ、エメラルドゴキブリバチ |
| 線形動物 | 線虫 |
| 植物 | シロイヌナズナ、モロコシ、イネ(Oryza sativa)、トマト、スイカ |
| 真正細菌 (バクテリア) |
鼻そ菌 ATCC 23344、類鼻そ菌 K96243 uid57733、枯草菌、Bradyrhizobium japonicum USDA 110 uid57599、大腸菌 K12、結核菌、ロドバクター・スフェロイデス 2.4.1 |
| 真核の菌類 | 酵母 |
対応データ
- FASTA
- FASTQ
- unaligned BAM
- tag-count format
- SAM
- BAM
動作環境
| 対応OS | Windows (Windows 7、8、10) Mac OS X (10.9~10.13)10.15、11以降では動作しません Linux (Red Hat Enterprise Linux 6.5まで, Debian GNU/Linux 9.0まで, Ubuntu 18.04 LTSまで) |
|---|
- * 最新バージョンを新規インストールする際の対応OSです。
- * OSアップデートについてはsupport@digital-biology.co.jpまでお問い合わせ下さい。
| CPU | 4core CPU 以上 |
|---|---|
| メモリ | 8GB RAM 以上 |
| ストレージ | 1TB以上の空き |
| ディスプレイ | 1024×768以上 |
- * 必要なディスクスペース、CPU、メモリは下記に依存します。
- * 詳しくは、下記を表記の上support@digital-biology.co.jpまでお問い合わせ下さい。
- 解析したいサンプル数
- アプリケーション
- リード数
- * インターネット接続必要
次世代シーケンスデータ解析ソフトウェア
「Strand NGS」のカタログを請求する
無料