よくあるご質問
近接ライゲーションテクノロジーDovetail Genomics®
すべて開く
Dovetail 受託サービスはどのような内容ですか?
「デノボアセンブリサービス」は、サンプルをお送りいただくだけで、ドラフトアセンブリ作成、近接ライゲーションライブラリ作成、スキャフォルディングまでを一括して行うことができます。「スキャフォルディングサービス」は、お手持ちのドラフトアセンブリとサンプルをお送りいただくことで、近接ライゲーションライブラリ作成以降の解析を行なうことができます。
Dovetail 受託サービスを利用することができない生物種はありますか?
生物種によっては、日本からDovetail社のあるアメリカまでの輸出・輸入が禁止、または制限がある場合があります。日本から輸出が可能か、アメリカに輸入が可能かを確認いたしますので、サンプルの生物種(学名)をお知らせください。この確認には数週間かかる場合があるため、あらかじめご了承ください。
Dovetail 倍数体にも対応できますか?
PacBio HiFi ロングリードとOmni-Cテクノロジーを組み合わせることで、多倍体のゲノムアセンブリにも対応が可能です。
サンプルにより、サブゲノムまたはハプロタイプの分解も可能ですのでご相談ください。
Dovetail サンプルはどのように輸送されますか?
国内輸送(お客様からトミーデジタルバイオロジー社まで)は、冷凍便でお送りください。弊社からDovetail社までは、ワールド・クウリアー社のサービスを使い冷凍輸送します。
Dovetail どれくらいの量のサンプルが必要ですか?
サンプルの生物種によって異なりますが、大まかな目安は以下の通りです。
動物 : 脳や肝臓など細胞密度の高い部位から、新鮮なうちに急速冷凍されたもの、200 mg程度。
植物 : 新芽や実生などできるだけ若い部位を、新鮮なうちに急速冷凍したもの、3 g 程度。
節足動物 : 胚や孵化したばかりの幼虫、初期の蛹などを、新鮮なうちに急速冷凍したもの、200 mg程度。
無脊椎動物 : 内臓(大型の動物の場合)、孵化したばかりの幼虫、精子、血液などを、新鮮なうちに急速冷凍したもの、500 mg程度。
サンプルのご準備に関する詳細は、別途資料がございますのでご連絡ください。
Dovetail QCはどのように行われますか?
Dovetail社で実施するクオリティチェックには以下のようなものがあり、お客様はチェックポイントごとの報告を受けることがあります。
1. ドラフトアセンブリ QC: ご提供いただいたドラフトアセンブリがHiRiseを使ったスキャフォルディングに使用できるかどうかをチェックします。
2. DNA QC : サンプルから抽出されたDNAの質、量、濃度がライブラリを作成するのに十分かどうかをチェックします。
3. ライブラリ QC : 近接ライゲーションライブラリが作成されたあと、少量のシークエンス(200M PE)を行ない、ドラフトアセンブリに対してマッピングします。このマッピング・アライメント情報を使用し、ライブラリの複雑性、リードペアの距離の分布、シグナルノイズ比を精査します。その結果、次に行なうディープシークエンスで、ゲノムカバレッジ 50x以上のデータを出力できると推測された場合、このクオリティチェックは通過となり、次のディープシークエンスに進みます。
4. スキャフォルドアセンブリ QC : HiRise パイプラインを使ってスキャフォルド解析を行った後、これまでの過去の結果を照らし合わせて、今回のアセンブリ結果が期待される結果だったかどうかが最終チェックされます。これが、お客様の元に納品される前の最後のQCです。
Dovetail データはどのように納品されますか?
基本的にはFTPによるダウンロードによりご提供いたします。ご要望に応じて、ハードディスクなどでデータを納品することも可能です。
ゲノムアセンブリ/スキャフォルディングサービス
・HiRiseによるゲノムアセンブリ結果(.fasta)
・概要レポート
・ドラフトスキャフォルドに推定されるミスジョイント情報
・ドラフトアセンブリ/スキャフォルドの位置情報
・アラインメントファイル(.bam)
・Omni-Cライブラリシークエンスデータ(.fasta)
+ TAD解析(可能な場合)
・コンタクトマトリクスファイル(.mcool, .hic)
・TADコール、CTCF結合サイト、A/Bコンパートメント、アイソコア位置(.bedpe)
・HiGlass用データと使用ガイド(.multires)
・概要レポート
エピジェネティクスサービス
・ライブラリシークエンスデータ(.fasta)
・アラインメントファイル(.bam)
・コンタクトマトリクスファイル(.mcool, .hic)
・ペアファイル(.pair)
・概要レポート
Dovetail 受託サービスではドラフトアセンブリはどのように作成しますか?
現在DovetailではPacBio HiFiロングリード × Hifiasmアセンブリの組み合わせを採用しています。
Dovetail 自分でドラフトアセンブリを作成する場合に、必要なデータ量はどれくらいですか?
illuminaの場合、目安として、2x150bpのペアエンドで最低60x カバレッジ、2x100bpの場合は90xカバレッジのデータ量が必要となります。
Dovetail HiRiseパイプラインに用いるドラフトアセンブリは、特別な方法で用意する必要がありますか?
いいえ。ドラフトアセンブリに使用するツールには、例えば、Discovar De Novo, SGA, Meraculous, Falconなど、様々なアセンブラがありますが、これでなければいけないという制限はありません。また、シークエンサーについても、HiRiseの必要条件(N50>100kb)に合致すれば、illuminaやPacBioなど制限はありません。しかし、最初のドラフトアセンブリの正確さが、次のスキャフォルディングの完成度に影響し、ひいてはアセンブリ全体の精度にも影響しますので、最初のドラフトアセンブリは、正確であればあるほど望ましいものになります。
Dovetail HiRiseに用いるドラフトアセンブリの必要最低条件は?
絶対的な必要最低条件ではありませんが目安として、
1)スキャフォルドまたはコンティグのN50が100kb以上
2)フローサイトメトリーなどで確認した推定ゲノムサイズの少なくとも75%がドラフトアセンブリに含まれていること
を推奨しています。より精度が高く、断片化の少ないドラフトアセンブリから、最終的には良いゲノムアセンブリ結果が得られるのはもちろんですが、これらはひとつの目安であり、サンプルのゲノムの特徴によって大きく変わります。
Dovetail N50は長いがエラーが多いドラフトアセンブリと、N50は短いがエラーが少ないドラフトアセンブリがある場合、どちらを解析に用いますか?
HiRiseの必要条件(N50>100kb)に合致する限り、N50は短くともエラーが少ないドラフトアセンブリを用いた方が、最終的には良いゲノムアセンブリ結果を得ることができます。
Dovetail 受託サービスで近接ライゲーションライブラリはどのように作成されますか?
Dovetail社では現在、近接ライゲーションライブラリの作成に通常、Omni-Cテクノロジーを用いています。サンプルのゲノム3Gbあたり1つの近接ライゲーションライブラリを作成します。
Dovetail 近接ライゲーションライブラリはどのようにシークエンスされますか?
Dovetail社では、HiSeq X シークエンサーを使用しています。
Dovetail 近接ライゲーションライブラリをシークエンスするために必要な条件は?
ペアエンドシークエンスで、最低100 bpあることが、HiRiseによる解析には必要です。より長いリード長(例:2x250bp)であれば、ハプロタイプの検出には効果的ですが、スキャフォルドの長さや質に大きく貢献するわけではありません。
Dovetail HiRiseアセンブリに必要な近接ライゲーションライブラリデータの条件は?
サンプルの生物種によって異なります。これに影響を及ぼす要因として以下のものがあります。
・ゲノム :サイズ、リピート配列の割合、GC含量、ヘテロ接合性
・近接ライゲーションライブラリ :複雑性、シグナルノイズ比、リードペアの距離分布(インプットDNAのサイズ)
・ドラフトアセンブリ : コンティグの長さと精度
ゲノムアセンブリに必要な近接ライゲーションライブラリのシークエンスデータは、ゲノム全体に均一に30x以上のカバレッジが必要です。
Dovetail 近接ライゲーションライブラリのデータは、たくさんあればあるほど、アセンブリ結果が良くなるものですか?
サンプルに依存しますが、多くとも100xカバレッジで十分な結果を得ることができます。Dovetail社では現在も、近接ライゲーションライブラリのデータ量がアセンブリの結果にどう影響するか調査していますので、これ以上データを増やして改善する場合もあるかもしれません。
Dovetail HiRiseでのスキャフォルドに必要なデータは何ですか?
HiRise パイプラインへは、ドラフトアセンブリ(N50>100kb)と、近接ライゲーションライブラリシークエンスがインプットデータとして必要です。
Dovetail 近接ライゲーションライブラリのリードペアはどのようにスキャフォルディングに使用しますか?
HiRise は、ドラフトアセンブリ上に推定される、偽結合箇所を切り離し、近接ライゲーションライブラリのリードペアで再結合し、より大きなサイズのスキャフォルド配列を作成することができます。そのためにHiRiseはプロジェクトごとに、作成した近接ライゲーションライブラリのインサートサイズ分布モデルを作成します。このモデルは、最初にマップした少量のシークエンスから作成されます。その後、そのモデルの推定値から、どの場所が切り離され、どの場所が再結合されるかを決定します。近接ライゲーションライブラリでは、2つのリードペア間距離が短いものは頻度が多く、長い距離をスパンするものは頻度が少なくなります。 HiRiseは多数の短いリードペアと少数の長いリードペアを使用して、アセンブリのコンティグまたはスキャフォルドを、様々な異なる向きと順序をテストして、その中で、想定されるインサートサイズ分布に最も適した向きと順序をそろえてつないだスキャフォルドを作成します。
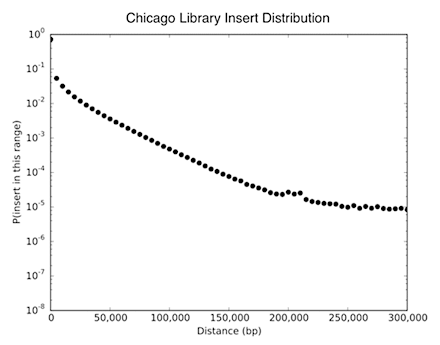
このグラフは典型的なChicagoライブラリのインサート距離の分布を示します。HiRiseによって導き出されたスキャフォルドの向きと順番が正しければ正しいほど、2本のスキャフォルドをつなぐリードペアの距離が、このインサート距離の分布モデルに一致するようになります。
Dovetail メイトペアやフォスミド、BACライブラリなどからのシークエンスデータを持っている場合、これらのデータをHiRiseに使用できますか?
これらの追加データは、現在のところHiRiseパイプラインに直接使用することはできません。しかしこれら追加データは、ドラフトアセンブリの結果を良くする可能性があり、ゲノムアセンブリ全体に見れば貢献します。また、オプティカルマッピングデータなどをお持ちの場合、HiRise後のスキャフォルド配列を改善するのに役に立つ可能性があります。さらに、カバレッジの低いBAC-endシークエンスや、RNA-seqデータなども、最終アセンブリ結果のバリデーションに用いることができると考えられます。
Dovetail 性染色体に興味がある場合は気をつけることは?
性染色体の配列、特に性染色体上の遺伝子を解析したい場合、2x以上のカバレッジが必要になります。
Dovetail ハプロタイプを分けて解析を行うことはできますか?
フェージング済みのドラフトアセンブリを用いることで、ハプロタイプごとのアセンブリを行なうことが可能ですが、その場合にはハプロタイプごとにHiRise解析費用が必要となります。
Dovetail ゲノムアセンブリの結果を良くするためにはどうすれば良いでしょうか?
ゲノムアセンブリの結果には複数の要因が複雑に関係しますが、HiRiseによるアセンブリの結果に最も影響が大きいものの一つは、インプットに使われるドラフトアセンブリのクオリティです。ドラフトアセンブリのコンティグ配列が長く、精度が高いほど、最終のゲノムアセンブリ結果も良くなります。
また、ドラフトアセンブリのクオリティは、リピートやヘテロ接合性、サイズなど、ゲノムの特徴に大きく依存します。またNGSリードの量や種類、使用したアセンブリアルゴリズムにもアセンブリ結果は大きく影響を受けます。
HiRiseアセンブリは、近接ライゲーションライブラリの配列データにも左右されます。近接ライゲーションデータで特にアセンブリに影響が大きいのは、ライブラリのゲノムカバレッジ均一性、リードペア間の距離の分布、シグナルノイズ比です。Dovetail社では、これらが最適な値を示すようなライブラリ調製を行なっています。近接ライゲーションライブラリ作成においては、ほとんどの要因はプロトコルで既に最適化されていますので、サンプルからどれだけ長くてきれいなDNAを抽出できるかが最も影響が大きくコントロール可能な要因の一つです。
Dovetail ゲノムアセンブリの結果はどのように評価することができますか?
標準的な参照ゲノム配列がある生物種の場合、アセンブル結果を参照配列と比較することでエラーの種類や頻度を算出できます。これまでDovetail社では、HiRiseアセンブリの精度を高めるため、参照配列がある生物種のアセンブリ結果を評価することで、プロトコルや解析アルゴリズムを最適化してきました。
参照配列が未知の生物種のアセンブリ結果を評価するためには、ゲノムアセンブリ結果と、進化的に最も近く既知のゲノム配列との間で、シンテニー(染色体上の遺伝子座の並び・順番)を比較する方法があります。もし近縁種で既知のゲノム配列が無い場合には、BAC-ends配列やトランスクリプトームデータを利用することが考えられます。BAC-endデータは数十kbから数百kbに及ぶ長鎖配列です。これらのデータをHiRiseアセンブリの結果配列にアラインすることで、予想インサートサイズやリードの向きを精査し、エラー頻度を算出することに用いることができます。
Dovetail TAD解析を行なうためにはどのようなゲノムアセンブリ結果が必要ですか?
Dovetail社では、以下のような条件を満たすゲノムアセンブリについて、TAD解析を行なう事が可能です。
・近接ライゲーションライブラリのリードペアの40%が1 Kbp以上の距離
・ゲノムサイズ < 10 Gb
・スキャフォルドサイズ < 400 Mbp
・ゲノムの40%以上がユニークにマッピング可能
・1Gbあたり100Mリードペアのカバレッジ
Dovetail どのキットを選択すればいいでしょうか?
目的のアプリケーションにより最適なアッセイが異なります。こちらの表をご参照ください。
| Dovetail™アッセイ | Omni-C | Micro-C | HiChIP |
|---|---|---|---|
| ゲノムトポロジー | ● | 〇 | |
| ゲノムスキャフォルディング | ● | ||
| 大規模構造変異 | ● | ||
| ジェノタイプ | ● | ||
| ハプロタイプ | ● | ||
| エンハンサープロモータ インタラクション | 〇 | 〇 | ● |
| タンパク質指向型クロマチン構造 | ● | ||
| ハイブリッドキャプチャの使用 | YES | YES |
●:Best fit ○:Good fit
Dovetail どのようなサンプルに用いることができますか?
Omni-Cキットは、ほ乳類の細胞、組織、血液、ほ乳類以外の動物組織、植物組織に用いることができます。
Micro-CとHiChIPについては、現在のところ、ほ乳類の細胞にのみ対応しています。その他の組織などへの対応は現在検証中です。
必要サンプル量や調製方法などの詳細については、プロトコル(ほ乳類用と、それ以外用があります)をご請求ください。
Dovetail HiChIP MNase キットではどのような抗体が選択できますか?
メーカーで検証済みの抗体リストがございます。こちらの表をご参照ください。
| DOVETAIL™ HICHIP VALIDATED ANTIBODY | SUPPLIER | PART NUMBER |
| IgG | Cell Signaling | 2729 |
| CTCF | Cell Signaling | 3418 |
| H3K4ac | Active Motif | 39381 |
| H3K4me3 | Cell Signaling | 9751 |
| H3K14ac | Cell Signaling | 7627 |
| H3K27ac | Cell Signaling | 8173 |
| H3K27me3 | Cell Signaling | 9733 |
| H3K36me3 | Cell Signaling | 4909 |
| Klf4 | Abcam | ab106629 |
| Nanog | Abcam | ab21624 |
| Oct4 | Cell Signaling | 2890 |
| Pol II | Abcam | ab26721 |
| Sox2 | Cell Signaling | 23064 |
Dovetail クロマチンの断片化はどのように行いますか?
Omni-Cの場合は、DNaseIを、Micro-CとHiChIPの場合はMicrococcal Nucleaseを断片化に用います。
Dovetail ブリッジアダプタはどのような配列ですか?
GGTTCGTCCATCgatc
(小文字が3′オーバーハング)となっています。
フルブリッジの配列は以下のようになります。
aGGTTCGTCCATCgatcGATGGACGAACCt
(ただし2×150 bp PEでシークエンシングすると、ほとんどの場合は、部分的にしかリードに入らないことにご注意ください)
Dovetail ライブラリのQCはどのように行えばいいでしょうか?
ちらのQCパイプラインを使用することができます。(https://github.com/dovetail-genomics/omni-c_qc)
Omni-C以外に、Micro-CとHiChIPでも同じパイプラインを使用することができます。
Dovetail 解析にはどれぐらいのカバレッジが必要ですか?
目的のアプリケーション(と解像度)、選択されたアッセイによって異なります。目安としては以下の通りです。
ゲノムアセンブリ – Omni-C: 30xカバレッジ(100 M PE reads/Gbp)
A/Bコンパートメント(解像度100 kbp) –
Omni-C: 2.7xカバレッジ(9 M PE reads/Gbp)
Micro-C: 2.5xカバレッジ(8 M PE reads/Gbp)
TAD(解像度10 kbp) –
Omni-C: 27xカバレッジ(90 M PE reads/Gbp)
Micro-C: 20xカバレッジ(67 M PE reads/Gbp)
Loop(解像度1 kbp) –
Omni-C: 160xカバレッジ(533 M PE reads/Gbp)
Micro-C: 80xカバレッジ(267 M PE reads/Gbp)
HiChIP –
Low Target abundance: 10xカバレッジ(33 M PE reads/Gbp)
High Target abundance: 40xカバレッジ(133 M PE reads/Gbp)
Dovetail サードパーティのNGSライブラリ調製キットを使用できますか
Omni-Cキットでは以下のキットを使用することが可能です。Micro-C, HiChIPキットについては現在検証中です。
– NEBNext® II DNA Library Prep Kit for Illumina®
– Kapa® Hyper Prep Kit
– Swift Accel-NGS® 2S Plus DNA Library Kit
Dovetail 下流のデータ解析にはどのようなツールを用いればよいでしょうか?
Dovetailで詳細なデータ解析ガイドをご用意しておりますので、こちらをご参照ください。
Omni-C: https://omni-c.readthedocs.io/en/latest/
Micro-C: https://micro-c.readthedocs.io/en/latest/
HiChIP MNase: https://hichip.readthedocs.io/en/latest/
Promoter Panel (Capture Micro-C): https://dovetail-capture.readthedocs.io/en/latest/
